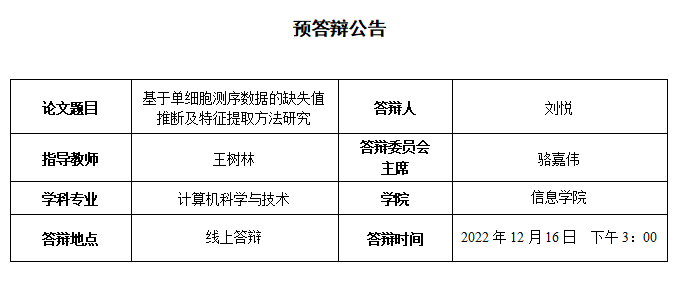
学位论文简介
自上个世纪以来,生命科学相关领域的发展日新月异,人类加快了探索人体结构和生命奥秘的步伐。细胞是人体结构的最基本单元,人体从一颗受精卵分化成为一个有120万亿细胞的成年人,这些细胞受同一组遗传信息控制,却在分化过程中各不相同。而正是这些差异,影响着人体的功能和健康。于是,相对于组织测序的概念,单细胞测序技术应运而生。而随着单细胞测序从低通量向高通量发展,产生了大量单细胞转录组学、表观基因组学、DNA甲基化等生物数据,这为以单细胞为切入点研究生物体的功能及健康提供良好契机。那么,如何对这些数据中包含的生物信号进行挖掘和分析是一项重大挑战。深入挖掘单细胞转录组学和表观基因组学中的表达信息,不仅对细胞间的通讯和胚胎发育等有着重要指导作用,还可以为精准医疗提供重要依据。然而,单细胞测序相关数据维度高且较稀疏,并且存在大量缺失事件,这对下游分析造成困扰。近年来,深度学习的发展使得基于深度模型的单细胞数据挖掘方法成为生物信息学领域的研究热点。本文以单细胞测序数据为基础,以深度算法为技术手段,针对单细胞测序数据的缺失值推断和特征提取任务进行研究。主要研究内容如下:
(1)由于扩增偏差、细胞周期效应等因素导致单细胞转录组测序数据的缺失事件,通过计算方法推断单细胞转录组测序数据缺失值是相对低成本和高效率的。本文提出了用于推断单细胞转录组数据缺失值的神经协同过滤算法,称为scNCF。首先,将单细胞转录组数据中的细胞和基因表示为独热编码,并通过不同维度的嵌入层将细胞和基因高维稀疏向量映射为低维稠密的隐向量。然后,将细胞和基因隐向量送入由多层感知机和矩阵分解构成的神经协同过滤模型,并预测未知表达值。神经协同过滤模型结合了矩阵分解的线性建模优势和多层感知机的非线性建模优势。不仅如此,scNCF还采用更适合缺失值推断任务的新的成对损失函数来优化模型。实验结果表明,scNCF的性能优于已有算法,而且具有适应于不同类型数据的可扩展性。
(2)对于极度稀疏的单细胞染色质可及性测序数据,由于技术限制等引起的缺失事件给聚类等下游分析造成困扰。尽管已经有十多种方法被提出专门用于推断单细胞转录组数据缺失事件,但尚未提出专门用于推断染色质可及性测序数据的方法。针对以上问题,本文选取近几年提出的较先进的单细胞转录组测序数据缺失值推断方法来推测单细胞染色质可及性测序数据中的缺失值。方法包括:MAGIC、SAVER、scImpute、deepImpute、PRIME、bayNorm和knn-smoothing,并通过几种下游分析全面对比几种方法的性能。具体分析包括聚类、亚群距离分析、轨迹分析等。不仅如此,为了评估这些方法的可扩展性,本文采用来自不同平台的规模不同的数据集,对比了他们的计算时间及在进一步缺失数据集上的性能。实验结果表明,多数方法恢复的峰值都提高了与元细胞的相关性。然而,在不同的下游分析中,不同的方法在不同数据集上的性能存在很大差异。总体而言,MAGIC和knn-smoothing表现最佳。
(3)单细胞多组学分析技术能够同时测量转录组学和表观基因组学图谱,为剖析细胞命运及异质性提供前所未有的机会。然而,多组学数据的稀疏、高维、近似二值化等特点严重阻碍了对其的分析。本文提出异构图跨组学注意力机制模型(scHGA)来提取单细胞的特征表示,这是一种基于单细胞异构图神经网络结合两种注意力机制来联合分析单细胞多组学数据的计算工具。具体来说,通过节点注意力机制获得基于元路径的细胞相似性图,再由跨组学注意力机制获得细胞特征表示,细胞特征表示可以捕获双组学细胞特征以进一步剖析细胞异质性。scHGA能够在具有不同异质特征的模拟数据及基于不同测序协议的真实数据上准确分析细胞类型。不仅如此,scHGA是既适用于多组学数据又可分析单组学数据的可定制化模型。scHGA是在单细胞多组学分析上图神经网络模型的成功探索,为分析和理解单细胞测序数据提供新的见解。
主要学术成果
[1] Yue Liu, Shulin Wang, Xiangxiang Zeng, et al. Are dropout imputation methods for scRNA-seq effective for scATAC-seq data? Briefings in Bioinformatics, 2022. (SCI 2区,CCF B类)
[2] Yue Liu, Shulin Wang, Junfeng Zhang. A heterogeneous graph cross-omics attention model for single-cell representation learning. International Conference on Bioinformatics & Biomedicine(BIBM 2022)(CCF B类)
[3] Yue Liu, Shulin Wang, Junfeng Zhang, et al. DMFMDA: Prediction of Microbe-Disease Associations Based on Deep Matrix Factorization Using Bayesian Personalized Ranking. IEEE/ACM Transactions on Computational Biology and Bioinformatics. 2020. (SCI 3区,CCF B类)
[4] Yue Liu, Shulin Wang, Junfeng Zhang, et al. A neural collaborative filtering method for identifying miRNA-disease associations. Neurocomputing. 2020. (SCI 2区)
[5] Yue Liu, Shulin Wang, Junfeng Zhang, et al. LncRNA-disease associations prediction based on neural network-based matrix factorization. IEEE Access. 2020. (SCI 2区)